2021年1月27日,中国科学院数学与系统科学研究院张世华研究员与中国科学院昆明动物研究所宿兵研究员、北京大学生命科学学院李程研究员团队合作,以“3D Genome of macaque fetal brain reveals evolutionary innovations during primate corticogenesis”为题,在国际顶尖学术期刊《细胞》在线发表最新研究成果。该研究构建了灵长类迄今最高分辨率的大脑三维(3D)基因组图谱,通过跨物种多组学分析和实验验证,揭示了三维基因组参与人类大脑发育的进化机制。

研究课题意象图。图中正在冥想的猴子表征灵长类大脑和智力的进化。
人类大脑起源于漫长的生命进化过程。哺乳动物从小鼠、猕猴到人类经历了上亿年的系统演化历史,其最显著的改变是大脑的认知功能,反映在脑容量的显著扩增和脑结构的高度精细化。人类进化过程中,哪些遗传改变造就了人类大脑,是国际科学界长期力图回答的重要科学问题。所有器官包括大脑的形成都是通过发育过程来实现的,人类独特的脑发育模式源于在进化中基因组积累的功能性突变。然而,如何将基因组中的序列差异与脑发育的调控改变之间建立因果联系,并解析其中的分子调控机制,是颇具挑战的研究问题。
在该研究中,研究人员通过学科交叉合作,开展了跨物种大脑发育三维基因组的研究。首先,利用Hi-C技术构建了中国猕猴胎脑的高分辨三维基因组图谱,这是目前包括人类在内的灵长类大脑分辨率最高的三维基因组图谱,达到了1.5 kb的分辨率,可以高精度地解析脑发育中基因组的空间组织方式。同时,还获得了猕猴胎脑的转录组图谱、染色质开放区图谱以及染色质锚定蛋白CTCF的分布图谱。综合这些猕猴胎脑的多组学图谱数据,研究人员首次构建了猕猴胎脑发育过程中的染色质精细空间构象,鉴定了包括染色质区室、染色质拓扑结构域(简称TAD)以及染色质环(简称Loop)等不同尺度的染色质结构,以及基因组在大脑发育中发挥重要作用的调控元件(如增强子等)。
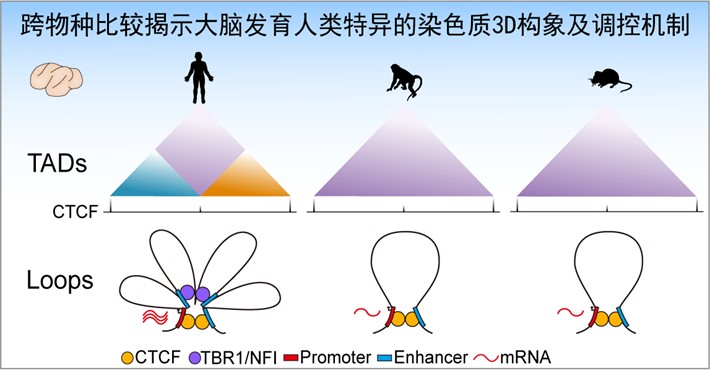
研究项目设计与研究结果图解(摘自《细胞》发表论文图解摘要)
通过与已发表的公共数据整合,研究人员接着进行了跨物种三维基因组的比较(人类、猕猴和小鼠),发现了多个具有人类特异染色质结构的基因组位点,包括499个人类特异TADs和1266个人类特异Loops。这些人类特异Loops显著富集增强子-增强子互作的调控模式,提示大脑发育在人类祖先中进化出更为精细的转录调控网络。重要的是,通过整合分析人脑发育的单细胞表达谱数据,发现这些人类特异Loops调控的基因在胎脑的SP(subplate)层显著表达,由此推测人类特异Loops对SP层的人类特异发育模式可能发挥重要作用。胎脑SP层是脑发育早期神经环路及神经可塑性形成的重要脑层,在人类进化过程中SP层出现了显著的扩张,其厚度可以达到皮层厚度的4倍左右。但由于在胎儿出生以后,该脑层逐渐消失,人们对其形成机制和功能了解较少。该研究结果首次为SP层在人类特异脑结构的发育和形成中的重要作用提供了证据。
这一研究成果首次产生了非人灵长类动物的高精度三维基因组学图谱资源,并利用大脑三维基因组的跨物种多组学分析,发现了人类特异的染色质结构和脑发育调控元件,为阐明人类大脑发育的进化机制提供了新思路和证据。该研究得到了中国科学院、国家自然科学基金委、科技部和云南省相关基金的资助。
张世华团队长期开展生物信息计算、数学建模、机器智能基础算法的研究,在计算三维基因组领域,还与合作者,针对单细胞三维基因组图谱数据,提出一种功能强大且稳健的环状轨迹重构工具CIRCLET。该方法考虑了染色体的多尺度结构特征,并无需指定起始细胞,以用于排序单细胞的周期阶段。轨迹重构有助于准确地刻画细胞周期过程染色质结构的绝缘强度和区室的动态变化,发现与动态子结构相关的重要调节基因等。为单细胞层面研究三维基因组图谱的动态调控提供了有力的工具(Advanced Science, 2019)。该成果入选2019年度中国生物信息学十大算法与工具。针对拓扑结构关联域识别问题,提出一个通用且高效的多尺度拓扑结构域识别方法MSTD,以从多种类型的三维基因组数据中鉴定多尺度的拓扑结构(Nucleic Acids Research, 2019)。
参考文献:
1. Xin Luo*, Yuting Liu*, Dachang Dang*, Xiaoyu Meng, Ting Hu, Tingting Li, Can Wang, Min Li, Xiechao He, Lanzhen Yan, Shihua Zhang#, Cheng Li#, Bing Su#. Comparative 3D genome analyses identify human-specific evolutionary changes during corticogenesis. Cell (2021), 184(3), Pages 723-740.e21. https://doi.org/10.1016/j.cell.2021.01.001.
2. Yusen Ye, Lin Gao#, Shihua Zhang#. Circular trajectory reconstruction uncovers cell-cycle progression and regulatory dynamics from single-cell Hi-C maps. Advanced Science (2019), 1900986.
Yusen Ye, Lin Gao#, Shihua Zhang#. MSTD: an efficient method for detecting multi-scale topological domains from symmetric and asymmetric 3D genomic maps. Nucleic Acids Research (2019), 47(11), e65.附件下载: