遗传变异指不同个体之间在DNA水平上的差异,是物种形成和生物进化的基础。1990年正式启动的人类基因组计划(Human Genome Project, HGP)与曼哈顿原子弹计划和阿波罗计划并称为三大科学计划。作为一项规模宏大,跨国跨学科的科学探索工程,HGP测定了组成人类染色体中所包含的30亿个碱基对组成的核苷酸序列,绘制人类基因组图谱。于2001年发布人类参考基因组的草图,被认为是HGP成功的里程碑事件,奠定了通过与参考基因组比较批量发现遗传变异的基础,并引发了测序的成本降低和吞吐量提升,以及用于组装和注释数据的数学和计算工具的发展。截止2021年,在庆祝人类基因组工作草图发布20周年时,经由DNA百科全书计划(ENCODE)以及大量的全基因组关联分析(GWAS)研究,发现在人类基因组非编码区域富集大量复杂疾病统计关联的遗传变异,以及进化中驱动复杂性状创新与重塑的适应性突变。这些序列变化引起的基因调控效果具有时间和空间依赖性,为系统破译这些位点的分子调控机制和功能效应带来了极大挑战。
为了应对这一挑战,中科院数学与系统研究院王勇团队与合作者发展了数学模型,即构建基因调控网络解析遗传变异的新方法论框架vPECA (Variants interpretation model by Paired Expression and Chromatin Accessibility data)。框架分为四个步骤。1)确定遗传变异富集的细胞类型或者特定的时间空间场景;2)测定该场景下的表观组-转录组等多组学数据;3)构建数学模型整合基因组-表观组-转录组-表型等数据,推断以调控元件为核心的基因调控网络模型。4)基于网络结构,系统解读复杂表型关联的遗传变异并分析其调控机制。vPECA的核心思想是重建遗传变异蕴藏信息和发挥功能的特定场景下的调控网络,通过具有调控活性的元件来建立遗传变异与下游基因和上游转录因子的因果关系。
基于vPECA框架,与中科院昆明动物所、斯坦福大学和西藏大学合作,对高原低氧环境适应的分子机制这一进化和遗传领域的核心科学问题开展研究。通过汉藏差异遗传变异的富集分析确定脐带静脉内皮细胞(HUVEC)和低氧场景;设计实验采集、测量、比较藏族适应型和汉族野生型两种HUVEC在低氧和常氧条件下不同时间节点上的多组学数据,包括基因组、转录组、染色质可及性(ATAC-seq)和染色质空间构象(Hi-C)数据;利用vPECA模型集成这些数据,系统解析藏族人群适应高原低氧环境的分子调控机制。揭示了EPAS1的基因表达由受选择和不受选择两类调控元件组合调控;位于增强子区域的功能位点通过削弱所在区域的染色质开放程度,进而下调EPAS1的表达,从而避免藏族人群在高原低氧环境下红细胞的过度增殖。构建了EPAS1基因的下游调控网络,探索了高原适应相关表型与分子调控层面的联系。成果发表在自然子刊Nature Communications,数学院博士生信晶雪为第一作者。
基于vPECA框架,与中科院基因组所、斯坦福大学、中科院上海营养与健康所和克莱姆森大学合作,对人脸面部特征相关遗传变异和调控机理这一遗传学核心科学问题开展研究。首先确定颅神经脊细胞(CNCC)为遗传变异发生功能的场景。CNCC起源于前脑,中脑和后脑的头部区域,迁移到发育中的颅面部区域,然后分化为多种细胞类型。 整个分层,迁移和分化过程受到严格调控,颅面发育过程中的异常会导致出生缺陷。通过构建一致性最优化模型,整合了多样本的匹配的基因表达和染色质可及性数据,重建了全基因组的CNCC人类调控网络hReg-CNCC, 预测了高质量的调控关系,揭示与神经板边界的发生,功能细化和迁移相关的上游,核心和下游转录因子的网络结构。 hReg-CNCC被系统用于注释脸部形态GWAS的遗传变异,揭示了对核心转录因子 ALX1的多个SNP的远程和组合调控,以及与面部距离和颅面骨稀有疾病的关联。此外,hReg-CNCC将进化中的DNA序列差异(例如超保守元件和人类加速进化区域)与基因表达和表型联系起来。 hReg-CNCC为颅面部特征等复杂表型研究提供了宝贵的资源,也强调了颅面部特征的遗传变异的解读需要放在胚胎早期发育的场景中进行。成果发表在自然旗下期刊Communications Biology,数学院博士生冯占营为第一作者。
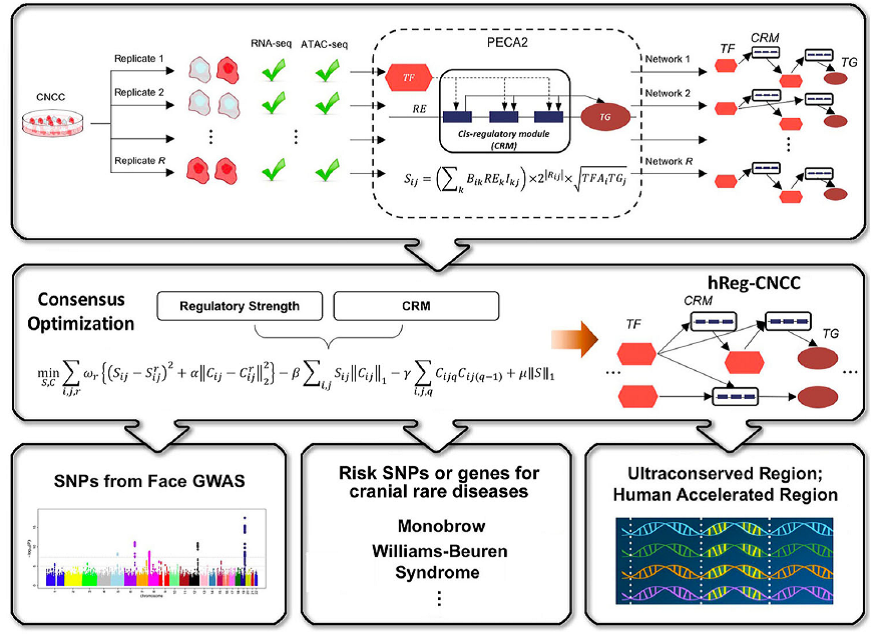
hReg-CNCC研究项目设计与研究结果
Z Feng, Z Duren, Z Xiong, S Wang, F Liu, WH Wong, Y Wang. hReg-CNCC reconstructs a regulatory network in human cranial neural crest cells and annotates variants in a developmental context. Communications Biology 4 (1), 1-16 (2021). https://www.nature.com/articles/s42003-021-01970-0
Jingxue Xin, Hui Zhang, Yaoxi He, Zhana Duren, Caijuan Bai, Lang Chen, Xin Luo, Dong-Sheng Yan, Chaoyu Zhang, Xiang Zhu, Qiuyue Yuan, Zhanying Feng, Chaoying Cui, Xuebin Qi, Wing Hung Wong, Yong Wang, Bing Su. Chromatin accessibility landscape and regulatory network of high-altitude hypoxia adaptation. Nature Communications. vol. 11: 1-20 (2020). https://www.nature.com/articles/s41467-020-18638-8
附件下载: