单细胞转录组学技术可以在单细胞分辨率下测量基因表达,为研究生物体发育过程、复杂疾病机制等生物学问题提供深刻见解。然而,单细胞RNA测序(Single-cell RNA sequencing, scRNA-seq)数据本身缺乏空间信息,而空间信息对于理解细胞分化、癌症微环境和大脑结构等方面起着至关重要的作用。近年来,空间转录组学(spatial transcriptomics, ST)技术的兴起使得研究人员可以在保留空间信息的情况下测量基因表达,为一些生物学问题的发现提供帮助,如涡虫的再生机制、人体心脏的发育、癌症微环境分析等研究问题。然而,ST技术仍然存在一些显著的局限性。以Spatial Transcriptomics和10x Visium为代表的技术,其数据具有分辨率低的特点,这意味着每个点由多个细胞组成。在高分辨率ST数据中,基于测序的数据,例如Slide-seqV2,虽然提高了空间分辨率,但数据质量较低;基于成像的数据,例如FISH,虽然信号质量较高,但测量的基因数量远少于基于测序的ST数据。
整合scRNA-seq数据与ST数据能够有效结合两种技术的优势。目前已有很多细胞类型反卷积的研究,它们利用scRNA-seq数据预测ST数据中每个点中各细胞类型的比例。然而,这些方法只能提供细胞群水平的空间位置预测,无法达到单细胞分辨率的空间定位。因此仍然迫切需要单细胞水平的空间定位方法,从而更精细地重构细胞的空间位置关系。
最近,中国科学院数学与系统科学研究院张世华研究员课题组在Cell Systems发表了题为《Spatial Transcriptomics-Aided Localization for Single-Cell Transcriptomics with STALocator》的文章,开发了空间转录组辅助的单细胞空间定位人工智能工具-STALocator。该方法的代码已经公开(https://github.com/zhanglabtools/STALocator)。

STALocator是一种通过与ST数据整合来空间定位scRNA-seq数据的方法(图1)。STALocator是一种基于深度学习的方法,它利用两个神经网络:一个将scRNA-seq数据与ST数据整合的整合网络,一个预测scRNA-seq数据的空间位置的定位网络。其中,整合网络采用改进的域迁移网络和切片Wasserstein距离来对齐scRNA-seq和ST数据,定位网络则使用空间信息监督的自编码器将ST数据的低维表示映射到特定空间位置。
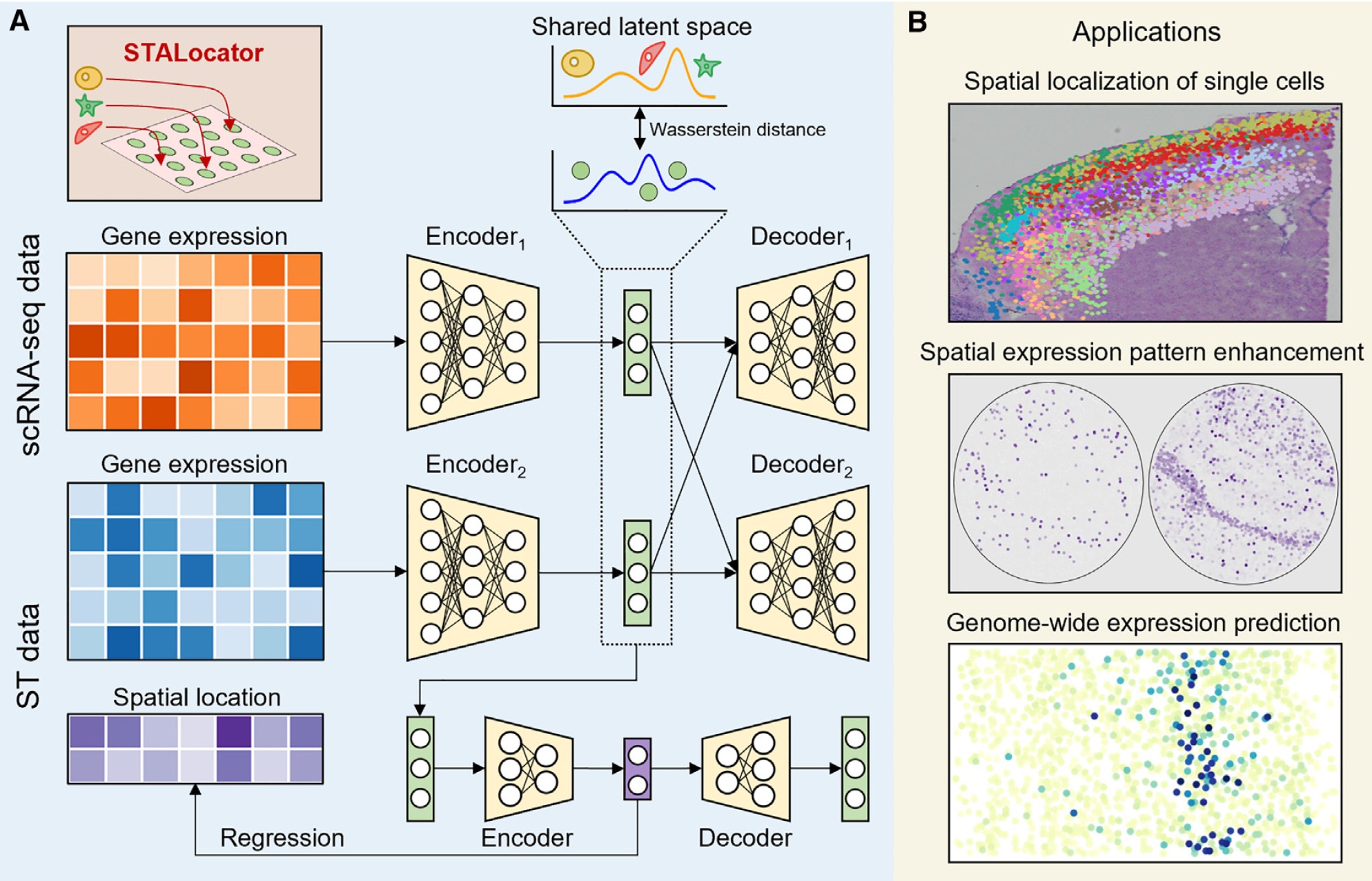
STALocator根据ST数据的分辨率采用不同的策略,使其适用于不同的生物场景。对于低分辨率ST数据,例如Spatial Transcriptomics和10x Visium,STALocator首先训练整合网络以获得消除批次效应的低维表示,然后训练定位网络来预测细胞的空间位置(图1B)。具有空间位置信息的单细胞数据可以看作是比原始ST数据更高分辨率的空间转录组数据。对于Slide-seqV2和FISH等分辨率接近scRNA-seq数据的高分辨率ST数据,STALocator仅训练整合网络,然后在训练过程中更新最优传输计划,最终获得从scRNA-seq数据到ST数据的全局最优传输计划,可用于增强ST数据。对于FISH数据,STALocator可以获取全基因组的ST数据,解决了可测量基因数量有限的限制。
张世华课题组于近年来分别发布了适应于不同空间转录组技术、不同生物组织的生物组织空间亚结构识别工具-STAGATE(https://www.nature.com/articles/s41467-022-29439-6),生物组织多切片空间转录组数据整合新工具-STAligner(https://www.nature.com/articles/s43588-023-00528-w),基于深度学习显著图的空间域特异可变基因识别方法-STAMarker(https://doi.org/10.1093/nar/gkad801),高密度空间转录组生成工具-STAGE(https://doi.org/10.1093/nar/gkae294),解码空间转录组学中的精细分辨率细胞群分布图-STASCAN(doi: https://doi.org/10.1101/2023.09.02.556029),人鼠跨物种全脑空间转录组数据对齐方法-BrainAlign(https://www.nature.com/articles/s41467-024-50608-2);并与合作者绘制了地中海涡虫再生过程中的三维空间转录组图谱-STAPR,系统鉴定了多个再生关键调控因子(https://www.nature.com/articles/s41467-023-39016-0)。
Publication:
Cell Systems Available online 3 February 2025, 101195
https://doi.org/10.1016/j.cels.2025.101195
Author:
Shihua Zhang
NCMIS, CEMS, RCSDS, Academy of Mathematics and Systems Science, Chinese Academy of Sciences, Beijing, China
School of Mathematical Sciences, University of Chinese Academy of Sciences, Beijing, China
Key Laboratory of Systems Health Science of Zhejiang Province, School of Life Science, Hangzhou Institute for Advanced Study, University of Chinese Academy of Sciences, Chinese Academy of Sciences, Hangzhou, China
Email:zsh@amss.ac.cn
附件下载: